doi: 10.56294/mw2024614
ORIGINAL
Evaluation of the usefulness of a predictive model for the diagnosis of COVID-19 in relation to the TR-PCR test and rapid test, in patients at the Univalle Sud Hospital
Evaluación de la utilidad de un modelo predictivo para el diagnóstico de COVID-19 en relación con la prueba de TR-PCR y prueba rápida, en pacientes del Hospital Univalle Sud
Rommer A Ortega-Martinez1 ![]() *, Miguel A Molina-Cardenas1
*, Miguel A Molina-Cardenas1 ![]() *, Carlos E. Román-Calvimontes2
*, Carlos E. Román-Calvimontes2 ![]() *
*
1Universidad Privada del Valle. Cochabamba, Bolivia.
2Universidad Mayor de San Simón, Cochabamba, Bolivia.
Citar como: Ortega-Martinez RA, Molina-Cardenas MA, Román-Calvimontes CE. Evaluation of the usefulness of a predictive model for the diagnosis of COVID-19 in relation to the TR-PCR test and rapid test, in patients at the Univalle Sud Hospital. Seminars in Medical Writing and Education. 2024; 3:614. https://doi.org/10.56294/mw2024614
Enviado: 12-12-2023 Revisado: 01-03-2024 Aceptado: 10-05-2024 Publicado: 11-05-2024
Editor: PhD.
Prof. Estela Morales Peralta ![]()
Autor para la correspondencia: Rommer A Ortega-Martinez *
ABSTRACT
Objective: early diagnosis for COVID-19 with rapid antigen test (PRA) and real-time polymerase chain reaction (TR-PCR), applying a predictive model.
Method: retro-prospective, observational, longitudinal study; in 2020, a PRA and TR-PCR indication score for COVID-19 was applied to 98 patients (age, fever, respiratory symptoms, chest X-ray, comorbidities, place of residence and work activity); > 4 indicated taking TR-PCR (χ2=0,531, p>0,05), area under the curve (AUC) of 0,5; 3 regression models were performed, the third was more effective (vital signs and clinical signs) to decide positivity; in 2023, 284 patients were included to validate the model.
Results: mean age 47,7±16,7, frequent symptoms with cough, fever and asthenia; mean systolic pressure 119,7±15,6 mmHg and diastolic 74,8±10,9 mmHg; TR-PCR with a sensitivity of 85 %, a validity rate of 49 %; positive predictive value of 48 %; negative predictive value 55 %, positive likelihood ratio 1,019; Cohen’s Kappa index, (κ=0,016, 95 % CI: -0,073 to 0,1).
Conclusions: molecular tests were contrasted with vital signs and clinical signs; We consider that our model can be useful, but its validation still needs to continue and be applied to more patients.
Keywords: COVID-19; Molecular Diagnostic Techniques; Predictive Models; Rapid Diagnostic Tests; Sensitivity; Specificity.
RESUMEN
Objetivo: el diagnóstico precoz, para COVID-19 con prueba rápida de antígeno (PRA) y la reacción en cadena polimerasa en tiempo real (TR-PCR), aplicando un modelo predictivo.
Método: estudio retro-prospectivo, observacional, longitudinal; en 2020 se aplicó a 98 pacientes, un score de indicación de PRA y TR-PCR para COVID-19 (edad, fiebre, sintomatología respiratoria, Rx de tórax, comorbilidades, lugar de residencia y actividad laboral); > de 4 indicaba tomar TR-PCR (χ2=0,531, p>0,05), área bajo la curva (ABC) de 0,5; se practicaron 3 modelos de regresión, el tercero fue más eficaz (signos vitales y clínica), para decidir positividad; el 2023 se incluyeron 284 pacientes para validar el modelo.
Resultados: media de edad 47,7±16,7, síntomas frecuentes con tos, fiebre y astenia; media de presión sistólica 119,7±15,6 mmHg y 74,8±10,9 mmHg diastólica; la TR-PCR con sensibilidad del 85 %, un índice de validez del 49 %; valor predictivo positivo de 48 %; valor predictivo negativo 55 %, razón de verosimilitud positiva de 1,019; índice Kappa de Cohen, (κ=0,016, IC 95 %: -0,073 a 0,1).
Conclusiones: las pruebas moleculares, se contrastaron con signos vitales, clínica; consideramos que nuestro modelo puede ser útil, empero aún se necesita continuar su validación y aplicarlo en más pacientes.
Palabras clave: COVID-19; Modelos Predictivos; Pruebas de Diagnóstico Rápido; Técnicas de Diagnóstico Molecular; Sensibilidad; Especificidad.
INTRODUCCIÓN
Un brote de una nueva neumonía, en Wuhan, China, el 2019, por el coronavirus (SARS-CoV-2, 2019-nCoV, COVID-19), se propagó rápidamente; donde el 81 % presentaban neumonía leve a moderada; el 2020, la tasa de mortalidad fue del 2,3 %;(1,2,3,4) la COVID-19, puede progresar a una enfermedad severa con alta mortalidad; el factor de gravedad son varones más de 65 años, y con mayor riesgo de requerir cuidados intensivos.(3,4)
Para su detección, se utilizan pruebas rápidas de antígenos (PRA) y el TR-PCR (reacción en cadena polimerasa de transcripción inversa); los ensayos con PRA iniciaron su comercialización en noviembre de 2020, y como complemento la TR-PCR; las PRA, detectan antígenos a partir de hisopos en las fosas nasales y devuelven resultados en minutos; tiene una sensibilidad de 40,2 al 74,1 % y una especificidad de 93 al 99,8 % en asintomáticos, que es inferior a la de TR-PCR (86 al 92 % y 99 %, respectivamente); sin embargo, las PRA son más baratas y fáciles de implementar a escala.(2,5)
Los hospitales deben contar con las PRA y TR-PCR, que permitan detectar a estos pacientes en etapa aguda, convalecencia o de seroconversión.(6) La OMS (Organización Mundial de la Salud) menciono que la prueba de elección para el diagnóstico fue la TR-PCR, realizadas en muestras de sangre y de vías respiratorias.(5,6)
El diagnóstico rápido y aislamiento de las personas portadoras de SARSCoV-2, son clave para romper la transmisión; la TR-PCR, es un proceso doloroso, presenta complicaciones, requiere personal capacitado y supone un riesgo de infección para la persona quién toma la muestra; de esta manera han aparecido varias opciones, como la de Kocagoz T et al, donde aplicaron al hisopo nasal, gárgaras y enjuagues bucales e identificaron al virus antes y después con 76 (66,7 %), 67 (58,8 %) y 101 (88,6 %) respectivamente.(7)
Una cohorte prospectiva del 2022, comparó la PRA con el índice de positividad de la TR-PCR (0 asintomático y 2 sintomático); la PRA mostro 93,4 % de sensibilidad a las 48 horas en sintomáticos y en comparación con el índice la sensibilidad, este subió de 62 a 79 % con la tercera toma.(8) Otro estudio realizado con la creación de 4 prototipos por 3D de hisopos nasofaríngeos, se encontró un índice de concordancia de las del 80 % (K=0,85-0,89) con pruebas TR-PCR.(9)
Por otro lado, hay muchos pacientes positivos, mientras que a los negativos no se puede descartar la posibilidad, generando los falsos negativos, lo que representa una amenaza para la comunidad; es así que hay otras opciones diagnósticas, como listas de cotejo, dispositivos en 3D, etc.; de esta manera, tras la invención de un score de predicción, tanto para la PRA y el TR-PCR, se creó un modelo predictivo convariables clínicas, epidemiológicas, constantes vitales, que se aplicó a personas que cursaron COVID-19; de tal manera, se pudo hacer una comparación si este modelo tiene utilidad o no para el diagnóstico del COVID-19, frente a la prueba rápida o TR-PCR.
MÉTODO
El presente trabajo surgió de un trabajo retrospectivo, realizado entre julio y septiembre del 2020, donde se creó y aplico en 98 pacientes, un score de indicación de TR-PCR para COVID-19 (edad, fiebre, sintomatología respiratoria, Rx de tórax, comorbilidades, lugar de residencia y actividad laboral), donde un valor > de 4 brindaba la indicación de tomar TR-PCR en relación con una PRA; sin embargo, se demostró que no hay relación para indicar TR-PCR o PRA (χ2=0,531, P>0,05), con un ABC de 0,5 (valor de p= >0,05) (ver archivo adicional 1 [https://drive.google.com/file/d/1kjvPcL_JBXbbohTr67MlNftNk4y66Wif/view]); posteriormente se practicaron 3 modelos de regresión logística binaria, donde el tercero permitió la creación de un modelo predictivo para decidir sobre la positividad de una TR-PCR o PRA.
Posteriormente, para evaluar la utilidad del modelo predictivo; se aplicaron dos etapas; con base en un estudio observacional, analítico, retrospectivo, transversal; en la primera etapa, entre enero y julio del 2023, se incluyeron 163 pacientes del hospital Univalle Sud; se realizó el análisis descriptivo para la validación del modelo predictivo; con cuadros resumen de información acerca de las covariables; por otro lado, se presentaron los resultados del modelo, contrastados con los de laboratorio; para los signos vitales, se presentaron diagramas de caja, discriminados según el resultado positivo o negativo para COVID-19.
El modelo se evaluó con un análisis relacional entre la salida del modelo y los resultados de TR-PCR; también se empleó para los resultados laboratoriales y las distintas covariables, mediante la prueba de independencia de χ2 o el test exacto de Fisher; posteriormente, se determinó el índice Kappa de Cohen (κ) para los casos válidos; adicionalmente, se determinaron la sensibilidad, especificidad, valor predictivo positivo, valor predictivo negativo y valores de razón de verosimilitud.(10,11,12)
En la segunda etapa, entre agosto y septiembre del 2023, tomando en cuenta la especificidad, como el valor predictivo negativo, como nulos, se decidió ampliar la muestra, especialmente considerando un mayor número de casos con TR-PCR negativo, por lo que de 121 pacientes, 163 fueron agregados, con un total de 284 casos; de igual manera se contrastó el resultado del modelo y los resultados laboratoriales mediante una prueba de independencia de χ2 y el cálculo del índice de concordancia de Cohen, como índice Kappa (κ); solo se ejecutó la prueba laboratorial de TR-PCR, debido a que la prueba rápida no se realizó en cerca del 50 % (n=143). El proceso se realizó en R v. 4.2.2, SPSS v. 19 y Excel v. 2018.
RESULTADOS
La media de edad fue de 47,7±16,7 años, más en mujeres que en varones, con 48±18 años y 47±16 años, respectivamente; (p>0,05). Los síntomas más frecuentes fueron tos, fiebre y astenia, con 55 %, 60 % y 69 % respectivamente; las frecuencias registradas presentan una relación significativa con los resultados del modelo para fiebre, astenia y disnea (p<0,05) (tabla 1).
|
Tabla 1. Frecuencia de casos para covariables del modelo, según resultados modelados para PCR |
||||
|
Características |
General N=2841 |
Negativo, N=421 |
Positivo, N=2421 |
Valor de p2 |
|
Tos |
|
|
|
0,2 |
|
Ausente |
74 (45 %) |
7 (33 %) |
67 (47 %) |
|
|
Presente |
89 (55 %) |
14 (67 %) |
75 (53 %) |
|
|
No registrado |
121 |
21 |
100 |
|
|
Fiebre |
|
|
|
0,002 |
|
Ausente |
65 (40 %) |
15 (71 %) |
50 (35 %) |
|
|
Presente |
98 (60 %) |
6 (29 %) |
92 (65 %) |
|
|
No registrado |
121 |
21 |
100 |
|
|
Astenia |
|
|
|
0,001 |
|
Ausente |
50 (31 %) |
0 (0 %) |
50 (35 %) |
|
|
Presente |
113 (69 %) |
21 (100 %) |
92 (65 %) |
|
|
No registrado |
121 |
21 |
100 |
|
|
Cefalea |
|
|
|
0,7 |
|
Ausente |
95 (58 %) |
13 (62 %) |
82 (58 %) |
|
|
Presente |
68 (42 %) |
8 (38 %) |
60 (42 %) |
|
|
No registrado |
121 |
21 |
100 |
|
|
Disnea |
|
|
|
0,038 |
|
Ausente |
90 (55 %) |
16 (76 %) |
74 (52 %) |
|
|
Presente |
73 (45 %) |
5 (24 %) |
68 (48 %) |
|
|
No registrado |
121 |
21 |
100 |
|
|
Mialgia |
|
|
|
0,4 |
|
Ausente |
84 (52 %) |
9 (43 %) |
75 (53 %) |
|
|
Presente |
79 (48 %) |
12 (57 %) |
67 (47 %) |
|
|
No registrado |
121 |
21 |
100 |
|
|
Características |
General N=2843 |
Negativo, N=423 |
Positivo, N=2423 |
Valor de p4 |
|
Presión sistólica (mmHg) |
120 (110, 130) |
110 (100, 120) |
120 (110, 130) |
<0,001 |
|
Presión diastólica (mmHg) |
80 (70, 80) |
70 (60, 70) |
80 (70, 80) |
<0,001 |
|
Presión arterial media (mmHg) |
91 (83, 97) |
80 (73, 90) |
92 (87, 97) |
<0,001 |
|
Frecuencia cardiaca |
90 (81, 105) |
87 (76, 100) |
90 (81, 105) |
0,4 |
|
Frecuencia respiratoria |
22 (18, 24) |
20 (20, 24) |
22 (18, 24) |
>0,9 |
|
Temperatura (°C) |
36,70 (36,40- 37,50) |
36,50 (36,30-36,70) |
36,70 (36,40-37,50) |
0,010 |
|
Saturación de oxígeno (%) |
89 (84, 92) |
86 (82, 93) |
90 (85, 92) |
0,14 |
|
Nota: 1n (%); 2Prueba de chi2 de Pearson; 3Mediana, rango intercuartil (RIQ); 4Prueba de suma de rango de Wilcoxon. |
||||
La media de la presión sistólica fue de 119,7±15,6 mmHg, siendo 74,8±10,9 mmHg la media para la presión diastólica; ni las presiones, ni las frecuencias cardíacas o respiratorias se comportan de manera normal (tabla 1), en términos de mediana (P50) y el rango intercuartil (IQR). Las presiones registradas y la temperatura tienen relación con el grupo de pertenencia al resultado del modelo de predicción de prueba TR-PCR positiva para COVID-19 (p<0,05). Por otro lado, puede apreciarse, que los estimadores para el grupo pronosticado como positivo para la prueba TR-PCR, son superiores en todos los signos vitales.
La distribución de valores registrados para los grupos según el resultado del modelo de predicción de casos de COVID-19 en la prueba TR-PCR, se presenta en la figura 1.
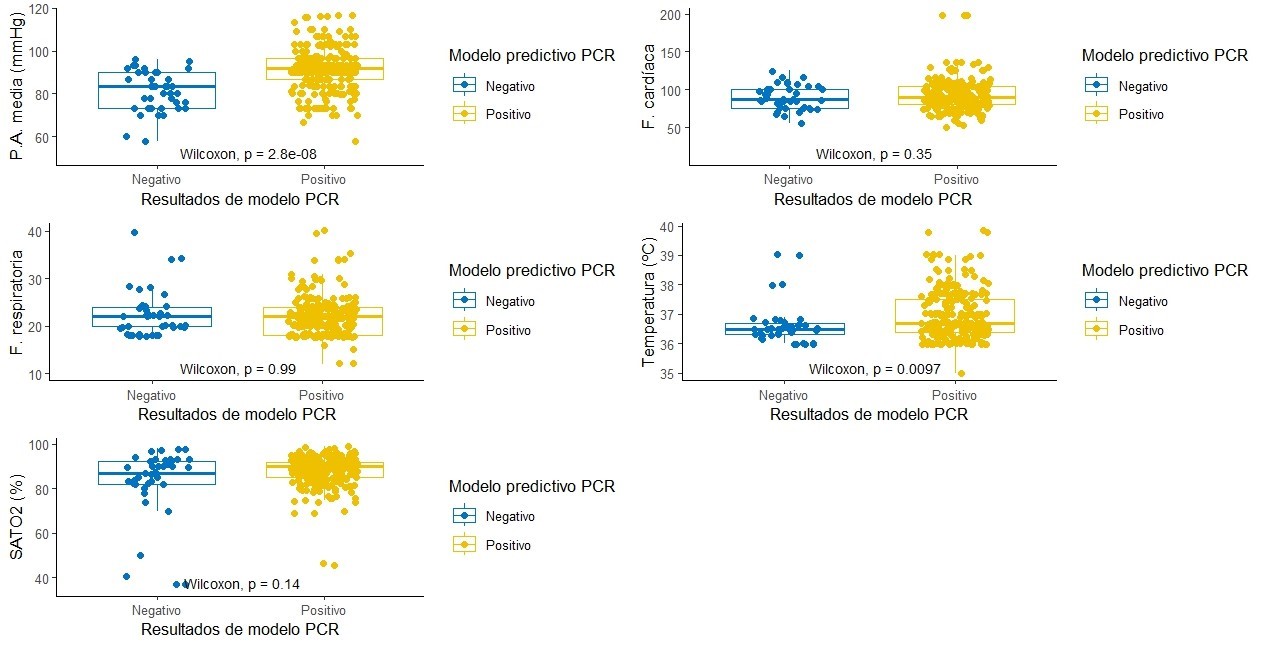
Figura 1. Resultados de medición de signos vitales según resultados de modelo predictivo para PCR.
Dentro el proceso de validación del modelo para la prueba rápida, la proporción de los casos en que no se realizó fue de 89 % (n=143), por lo que los valores que podrían ser contrastados entre el modelo de predicción para la prueba rápida, y los valores observados de la prueba rápida, carecen de relevancia; por lo indicado, se descartó un proceso adicional para la validación.
Cerca del 16 % de los casos no fueron analizados para TR-PCR (n=45); el resultado observado de casos positivos fue de 113; estos fueron catalogados por el modelo en 85 % de las ocasiones (n=96); sin embargo, el modelo no fue exitoso para 17 % de los casos de TR-PCR negativo (n=21); la prueba de independencia de χ2, indica que no existe relación entre las los niveles observados y modelados (p>0,05) (tabla 2).
|
Tabla 2. Matriz de contingencia para resultados del modelo PCR y resultados observados en laboratorio PCR |
|||||
|
Características |
General, N=2841 |
Negativo, N=1261 |
No realizado, N=451 |
Positivo, N=1131 |
Valor de p2 |
|
Resultado modelo PCR |
|
|
|
|
0,4 |
|
Negativo |
42 (15 %) |
21 (17 %) |
4 (8,9 %) |
17 (15 %) |
|
|
Positivo |
242 (85 %) |
105 (83 %) |
41 (91 %) |
96 (85 %) |
|
|
Nota: 1n (%); 2Prueba de chi2 de Pearson. |
|||||
Gráficamente, puede apreciarse mejor la proporción de aciertos y desaciertos del modelo, en relación con los resultados laboratoriales (figura 2); el número de casos en los que no se realizó el análisis laboratorial, podría estar generando el azar de los resultados. Descartando los casos en que no se realizó la prueba de TR-PCR, la matriz de contingencia demuestra un índice de validez o exactitud de 49 %; la sensibilidad es elevada (85 %), aunque el valor predictivo positivo es moderado (48 %); por su lado, la razón de verosimilitud positiva es de 1,019; sin embargo, tanto la especificidad (17 %), como el valor predictivo negativo (55 %); reflejan la debilidad del modelo para el caso de los verdaderos negativos.
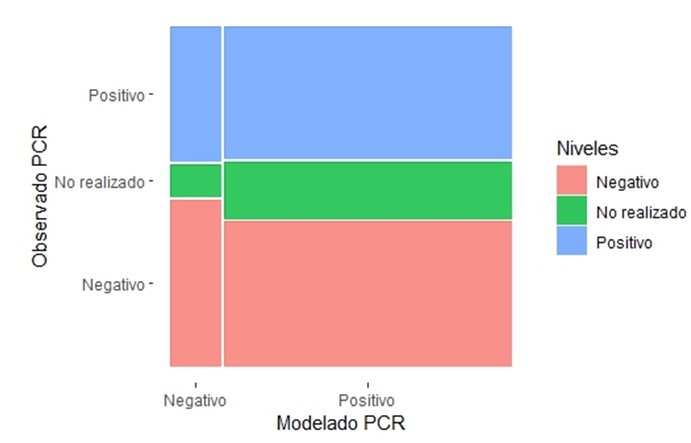
Figura 2. Diagrama de mosaico entre resultados del modelo y resultados de prueba PCR
El valor del índice Kappa de Cohen, indica una insignificante concordancia entre los valores de modelación y resultados laboratoriales, debido a la influencia de la debilidad del modelo para clasificar los valores verdaderos negativos (κ=0,016, IC 95 %: -0,073 a 0,1).
DISCUSIÓN
El impacto de la pandemia de COVID-19, reflejó la falta de insumos para enfrentar un infortunio de esta magnitud y una conmoción económica; dejando a relucir este inconveniente en contra del diagnóstico precoz y tratamiento idóneo; por otro lado, demostró la fragilidad del ser; de esta manera este estudio expuso una media de edad con 47,7±16,7 años, sin diferencia significativa entre varones y mujeres (valor de p>0,05); muy parecido al estudio de Soni A. et al donde presentaron una media de 37,4 años (8) y otros tomaron en cuenta grupos etarios variados con rangos de 2 a 90 años o la mayoría más de 18 años.(7,9)
Los síntomas son respiratorios como tos, fiebre y astenia, con 55 %, 60 % y 69 %, respectivamente, como el estudio de Achenbach C. et al, que mostro más del 40 % con tos, fiebre y cefalea.(10,11,12,13) Con base en los signos vitales en el score de predicción y en el tercer modelo de predicción, se ha observado que la presión arterial y la temperatura tienen relación con el grupo de predicción para la prueba positiva de TR-PCR (valor de p=<0,001); de igual forma un estudio con una estructura similar al nuestro, de Youssef Ali Amer A. et al en Bélgica, con la creación de cinco modelos predictivos, utilizando los signos vitales, determinaron una predicción de estado de salud para COVID-19, con el 8 %, 4,8 %, and 17,8 % para la frecuencia cardiaca, saturación de oxígeno y frecuencia respiratoria respectivamente, disímil a nuestros hallazgos.(14)
Debido a una reducida ejecución de PRA, el proceso de validación para este modelo sólo consideró el test exacto de Fisher; de acuerdo con la prueba, los resultados del modelo son independientes a los resultados laboratoriales (p>0,05); no obstante, en un estudio realizado por Callahan C. et al, donde refleja la utilidad de hisopos nasofaríngeos como prueba rápida de antígeno en 3D en comparación con un índice de positividad de la TR-PCR; la PRA mostro 93,4 % de sensibilidad a las 48 horas en sintomáticos y en comparación con el índice la sensibilidad subió a 79 % con la tercera toma;(8,13) otro estudio con la creación de 4 prototipos por 3D de hisopos nasofaringeso, encontró un índice de concordancia del 80 % (K=0,85-0,89) con pruebas TR-PCR,(9,13) esto expresa una disimilitud con nuestro medio por el índice de Kappa bajo, ya que la tendencia es solicitar más pruebas de TR-PCR.
Con respecto a las pruebas moleculares, las mismas han presentado una evolución tecnológica, como en el estudio de Bos D. et al donde revelaron que una prueba rápida de TR-PCR COVID-19 (miDiagnostics ®), alcanzo una sensibilidad del 95,5 % al 98,6 %;(14,15) de igual manera, se compararon al hisopo nasal clásico, gárgaras y enjuagues bucales como pruebas moleculares, antes y después, mostrando un 76 (66,7 %), 67 (58,8 %) y 101 (88,6 %) de sensibilidad respectivamente;(7,14) en nuestro estudio, el modelo catalogó adecuadamente a 85 % de los casos, lo que se refleja en valores elevados de sensibilidad, razón de verosimilitud y valor predictivo positivo; también es razonable el índice de validez o exactitud general (81,4 %); sin embargo, la especificidad y el valor predictivo negativo son nulos.
Otro estudio realizado por Joshi R. et al, donde utilizó un hemograma con base a un modelo predictivo y confirmaron con TR-PCR (+) a 33 pacientes y 357 pacientes (-), en servicios de emergencias; demostró una sensibilidad del 93 %, con un ahorro de más del 30 % en comparación con la TR-PCR ; indican que de 1000 pacientes, se evitaría pedir TR-PCR al 60 %; en el caso nuestro, cuando el paciente es sintomático, el modelo es capaz de coincidir con el laboratorio en por lo menos 8 de cada 10 casos.(15,16)
La inteligencia artificial, en comparación con la TAC de tórax y la clínica del paciente demostró un ABC de 0,92, identificando los TR-PCR (+) a un 68 %, demostrado por Mei X. et al en China.(17) Otro estudio el 2023, liderado por Alemi F. et al, donde aplicaron aspectos clínicos, identificados por modelos predictivos, además de prueba rápida en domicilio, obteniendo una sensibilidad del 69 % y especificidad del 98 % para tener una TR-PCR (+),(18) no así en el nuestro, que identifico un 85 % de sensibilidad, sobre todo por la cantidad de resultados negativos; en contraste, otro estudio con el uso de un analizador de diagnóstico para PCR, de hibridación selectiva (DASH, con la sigla en inglés), que además utilizo la prueba rápida, reflejo una sensibilidad del 96 % y especificidad del 98 %; un VPP de 85 % y 99 % de VPN.(13,18)
Existe un nulo acuerdo entre los resultados del modelo y las observaciones, reflejado en el valor del índice Kappa de Cohen (κ=0,016), en contraste con otros estudios, como el de Callahan CJ. et al, donde por medio de hisopos hechos por 3D, demostró una concordancia de κ=0,85-0,89;(9,18) los resultados indican la necesidad de incrementar la muestra, considerando valores negativos de TR-PCR; el modelo clasificó adecuadamente al 85 % de casos positivos, lo que es corroborado por sus valores de sensibilidad y razón de verosimilitud positiva; este es débil en la clasificación de casos verdaderos negativos, reflejado en una reducida especificidad (17 %) y un moderado valor predictivo negativo (VPN).
Siguiendo la línea de pruebas versátiles, Zowawi HM et al, el 2021 desarrollan un dispositivo portatil (Sistema Biomeme SARS-CoV-2®), por medio de hisopos nasales y nasofaringeso, brindan un resultado molecular en 80 minutos, con un porcentaje de similitud para positivo (99 %) y para negativo (97,8 %), aunque en nuestro estudio llega a un 85 %, aun viene a ser un sistema pertinente,(1,17) desde otro punto de vista, el 2023 se evaluó una PRA ultra rápida en comparación con la TR-PCR (Diagnovir ®), reflejando una sensibilidad del 100 y el 96,97 % respectivamente, no así en nuestro estudio que mostro el 85 %; la especificidad con 94 y 99,2 %, diferentes al nuestro, exponiendo que nuestro modelo aun necesitas más pruebas y validaciones.(2,18)
CONCLUSIONES
La necesidad de contar con pruebas accesibles, económicas, de fácil uso y confiables, han permitido la prueba de varias técnicas, scores, hisopos en 3D y en muchos casos aplicando la inteligencia artificial, pero hasta el momento ninguno recomendado, por lo que el modelo en este estudio, con una especificidad nula, sirve para considerarlo en el diagnostico por su sensibilidad; sin embargo, es verídico que podrían necesitarse más pacientes, combinando adecuadamente los resultados positivos y negativos para TR-PCR y evaluar si es posible continuar solicitando PRA, ya que últimamente se ha visto común solicitar únicamente pruebas moleculares; desde otra óptica, es vital aplicar este modelo en otras instituciones buscando pacientes más heterogéneos.
REFERENCIAS BIBLIOGRÁFICAS
1. Callahan CJ, Lee R, Zulauf KE, Tamburello L, Smith KP, Previtera J, et al. Open Development and Clinical Validation of Multiple 3D-Printed Nasopharyngeal Collection Swabs: Rapid Resolution of a Critical COVID-19 Testing Bottleneck. J Clin Microbiol [Internet]. 2020 Aug 1 [cited 2024 Nov 25];58(8). Available from: https://pubmed.ncbi.nlm.nih.gov/32393482/
2. Zowawi HM, Alenazi TH, AlOmaim WS, Wazzan A, Alsufayan A, Hasanain RA, et al. Portable RT-PCR System: a Rapid and Scalable Diagnostic Tool for COVID-19 Testing. J Clin Microbiol [Internet]. 2021 May 1 [cited 2024 Dec 4];59(5). Available from: https://pubmed.ncbi.nlm.nih.gov/33674285/
3. Seymen AA, Gulten E, Ozgur E, Ortaç B, Akdemir I, Cinar G, et al. Clinical evaluation of DIAGNOVIR SARS-CoV-2 ultra-rapid antigen test performance compared to PCR-based testing. Sci Rep [Internet]. 2023 Dec 1 [cited 2024 Dec 4];13(1). Available from: https://pubmed.ncbi.nlm.nih.gov/36932107/
4. Pan Y, Li X, Yang G, Fan J, Tang Y, Zhao J, et al. Serological immunochromatographic approach in diagnosis with SARS-CoV-2 infected COVID-19 patients. J Infect [Internet]. 2020 Jul 1 [cited 2024 Nov 24];81(1):e28–32. Available from: https://pubmed.ncbi.nlm.nih.gov/32283141/
5. Brodin P. Immune determinants of COVID-19 disease presentation and severity. Nature Medicine 2021 27:1 [Internet]. 2021 Jan 13 [cited 2024 Nov 24];27(1):28–33. Available from: https://www.nature.com/articles/s41591-020-01202-8
6. Wu S, Archuleta S, Ming LS, Somani J, Chye QS, Fisher D. Serial antigen rapid testing in staff of a large acute hospital. Lancet Infect Dis [Internet]. 2022 Jan 1 [cited 2024 Nov 24];22(1):14–5. Available from: https://pubmed.ncbi.nlm.nih.gov/34883064/
7. Chen J, Niu Z, Li H, Tang D, Zhang P. Analysis of Nucleic Acid and Antibody Detection Results for SARS-CoV-2 Infection. Arch Iran Med [Internet]. 2021 May 1 [cited 2024 Nov 24];24(5):427–33. Available from: https://pubmed.ncbi.nlm.nih.gov/34196209/
8. Kocagoz T, Can O, Yurttutan Uyar N, Aksoy E, Polat T, Cankaya D, et al. Simple concentration method enables the use of gargle and mouthwash instead of nasopharyngeal swab sampling for the diagnosis of COVID-19 by PCR. Eur J Clin Microbiol Infect Dis [Internet]. 2021 Dec 1 [cited 2024 Nov 24];40(12):2617–22. Available from: https://pubmed.ncbi.nlm.nih.gov/34448088/
9. Soni A, Herbert C, Lin H, Yan Y, Pretz C, Stamegna P, et al. Performance of Rapid Antigen Tests to Detect Symptomatic and Asymptomatic SARS-CoV-2 Infection : A Prospective Cohort Study. Ann Intern Med [Internet]. 2023 Jul 1 [cited 2024 Nov 24];176(7):975–82. Available from: https://pubmed.ncbi.nlm.nih.gov/37399548/
10. SOLUTION: Essential medical statistics 2nd ed b kirkwood j sterne wiley blackwell 2003 ww - Studypool [Internet]. [cited 2024 Nov 22]. Available from: https://www.studypool.com/documents/16816205/essential-medical-statistics-2nd-ed-b-kirkwood-j-sterne-wiley-blackwell-2003-ww
11. Roman J _Francisco. Guía Práctica del Curso de Bioestadística Aplicada a las Ciencias de la Salud [Internet]. [cited 2024 Nov 22]. Available from: https://www.academia.edu/10906749/Gu%C3%ADa_Pr%C3%A1ctica_del_Curso_de_Bioestad%C3%ADstica_Aplicada_a_las_Ciencias_de_la_Salud
12. Montgomery D, Runger GC. Probabilidad y estadística para ingenieros - Montgomery y Runger - III Ed. Probabilidad y Estadística aplicadas a la Ingeniería [Internet]. 2003 [cited 2024 Nov 22];486. Available from: https://www.academia.edu/10272859/Probabilidad_y_estad%C3%ADstica_para_ingenieros_Montgomery_y_Runger_III_Ed
13. Achenbach CJ, Caputo M, Hawkins C, Balmert LC, Qi C, Odorisio J, et al. Clinical evaluation of the Diagnostic Analyzer for Selective Hybridization (DASH): A point-of-care PCR test for rapid detection of SARS-CoV-2 infection. PLoS One [Internet]. 2022 Jun 1 [cited 2024 Nov 26];17(6):e0270060. Available from: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0270060
14. Amer AYA, Wouters F, Vranken J, Dreesen P, Boer D de K de, Rosmalen F van, et al. Vital Signs Prediction for COVID-19 Patients in ICU. Sensors (Basel) [Internet]. 2021 Dec 1 [cited 2024 Nov 26];21(23):8131. Available from: https://pmc.ncbi.nlm.nih.gov/articles/PMC8662454/
15. Bos DAG, Lagrou K, Verbakel JY. Prospective Performance Evaluation of the miDiagnostics COVID-19 PCR Test for Rapid SARS-CoV-2 Detection on Nasopharyngeal Swabs. J Clin Microbiol [Internet]. 2023 May 1 [cited 2024 Nov 26];61(5). Available from: https://pubmed.ncbi.nlm.nih.gov/37093001/
16. Joshi RP, Pejaver V, Hammarlund NE, Sung H, Lee SK, Furmanchuk A, et al. A predictive tool for identification of SARS-CoV-2 PCR-negative emergency department patients using routine test results. J Clin Virol [Internet]. 2020 Aug 1 [cited 2024 Nov 26];129. Available from: https://pubmed.ncbi.nlm.nih.gov/32544861/
17. Mei X, Lee HC, Diao K yue, Huang M, Lin B, Liu C, et al. Artificial intelligence-enabled rapid diagnosis of patients with COVID-19. Nat Med [Internet]. 2020 Aug 1 [cited 2024 Dec 1];26(8):1224–8. Available from: https://pubmed.ncbi.nlm.nih.gov/32427924/
18. Alemi F, Vang J, Bagais WH, Guralnik E, Wojtusiak J, Moeller FG, et al. Combined Symptom Screening and At-Home Tests for COVID-19. Qual Manag Health Care [Internet]. 2023 Jan 1 [cited 2024 Dec 1];32(Suppl 1):S11–20. Available from: https://pubmed.ncbi.nlm.nih.gov/36579704/
AGRADECIMIENTOS
Se agradece a la Universidad Privada del Valle, Cochabamba, Bolivia, por el apoyo y asesoría en el análisis estadístico de la información.
FINANCIACIÓN
Los autores no recibieron financiación para el desarrollo de la presente investigación.
CONFLICTO DE INTERESES
Los autores declaran que no existe conflicto de intereses.
CONTRIBUCIÓN DE AUTORÍA
Conceptualización: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Curación de datos: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Análisis formal: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Investigación: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Metodología: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Administración del proyecto: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Recursos: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Software: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Supervisión: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Validación: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Visualización: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Redacción – borrador original: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.
Redacción – revisión y edición: Rommer A Ortega-Martinez, Miguel A Molina-Cardenas, Carlos E. Román-Calvimontes.